Figure 1.1: Rappresentazione schematica dei processi di perdita di energia per una particella carica pesante.

Figure 1.1: Rappresentazione schematica dei processi di perdita di energia per una particella carica pesante.
|
In fig 1.2 sono illustrati i valori di Zeff in funzione della energia specifica (energia per unitá di massa atomica; solitamente espressa in MeV/u) della particella proiettile.
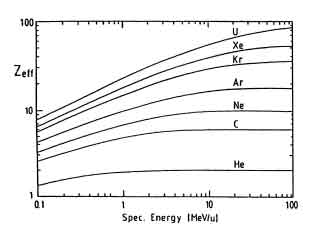
Figure 1.2: Carica efficace secondo la formula di Barkas in funzione della energia specifica.
|
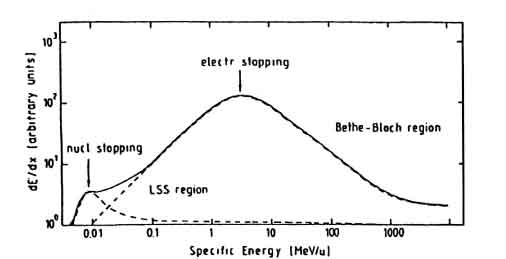
In fig. 1.3 vengono comparati valori teorici (Bethe-Bloch) ed empirici della perdita di energia (potere frenante massico) in funzione della energia specifica e dalla particella incidente:
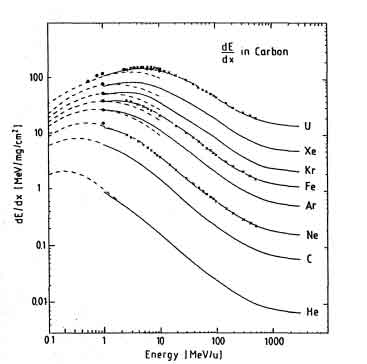
Figure 1.3: Comparazione tra la perdita di energia misurata con quella teorica in funzione della energia specifica.
|
Essendo dE/dx un valore "medio" anche R risulta avere una dispersione s che definisce il parametro di straggling a:=�{2s}. In pratica viene definito range medio la distanza per la quale il numero di particelle iniziale si é ridotto del 50% e straggling la FWHM (Full Width Half Maximum) della gaussiana che ne rappresenta la dispersione come si puó vedere in fig. 1.4 dove sono rappresentate due curve: la curva integrale che descrive l'andamento del numero di particelle ancora presenti ad una profonditá x e la curva differenziale che mostra il numero di particelle il cui cammino é pari alla profonditá x in ascissa.
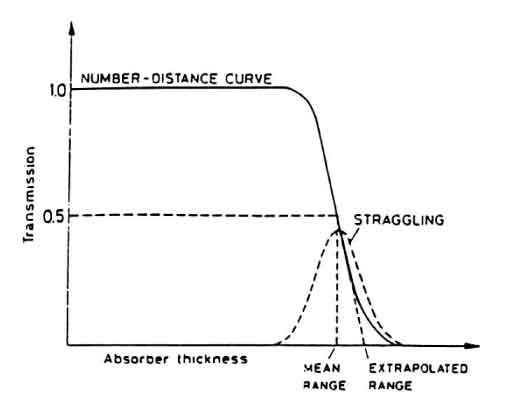
Figure 1.4: Curva integrale e differenziale dei percorsi di particelle cariche pesanti nella materia.
|
|
|
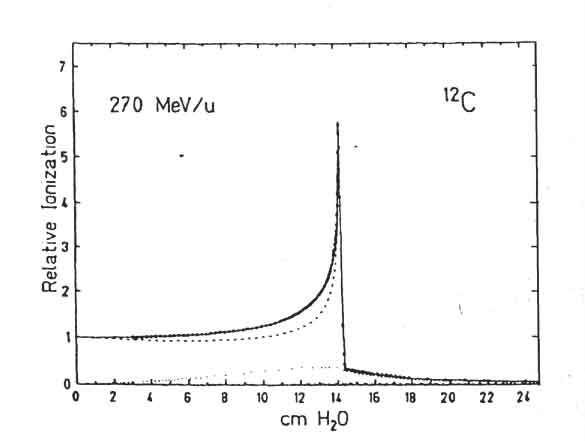
Figure 1.5: Densitá di ionizzazione in funzione della profonditá. La curva tratteggiata rappresenta il contributo dei frammenti nucleari.
Gli elettroni secondari
hanno quindi scattering elastici ed anelastici. Gli scatter elastici non
dissipano praticamente energia ma determinano la distribuzione angolare degli
elettroni; quelli anelastici vanno distinti in energia: A basse energie ( E
< 20 eV ) gli elettroni secondari non generano praticamente ionizzazione
ma si limitano ad eccitare gli atomi del materiale attraversato. A maggiori
energie domina invece la ionizzazione e tale rilascio di "Calling ghostscript
to convert zImages/fig3.EPSF to zImages/fig3.EPSF.gif , please wait ..." "Calling
ghostscript to convert zImages/fig4.EPSF to zImages/fig4.EPSF.gif , please wait
..." energia ha un massimo tra i 70 ed i 200 keV, per questo motivo elettroni
altamente energetici diventano maggiormente reattivi a livello biologico (dove
contano le ionizzazioni) quando vengono rallentati ad energie di poche centinaia
di eV. L'energia media trasferita negli eventi di ionizzazione é largamente
indipendente dalla energia degli elettroni e vale tra i 15 e 20 eV.
La combinazione del trasporto e del rilascio di energia degli elettroni consente
a codici Montecarlo di calcolare con ottima
verosimiglianza le tracce lasciate da ioni nella materia. Un esempio di tali
tracce é illustrato in fig. 1.7 .
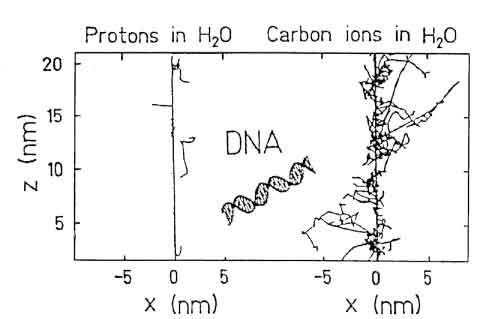
Figure 1.7: Comparazione tra le tracce di un protone e di uno ione carbonio entrambi con energia specifica di 1 MeV/u, in mezzo é rappresentata schematicamente la struttura del DNA: lo ione carbonio produce una maggiore densitá di ionizzazione causando danni piú elevati al DNA.
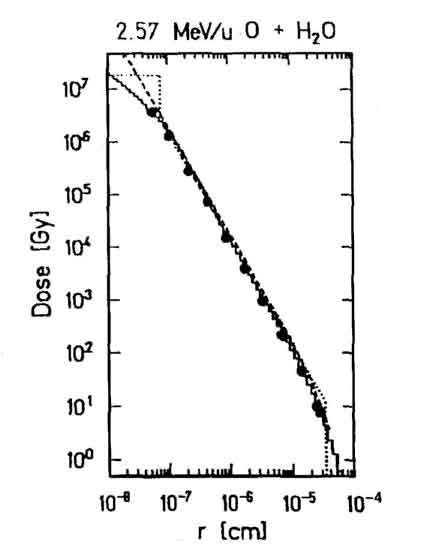
Figure 1.8: Distribuzione radiale della dose attorno alla traccia di una particella: la dose decresce come 1/r2 su vari ordini di grandezza della distanza radiale.
|
|
|
|
|
|
|
|
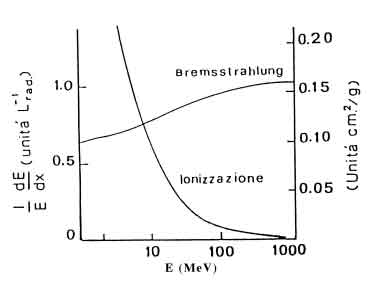
Figure 1.9: Contributi delle due modalitá di perdita di energia di elettroni in funzione della loro energia.
|
|
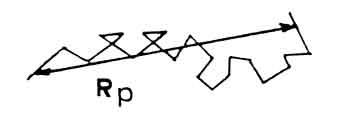
Figure 1.10: Traettoria di un elettrone all'interno della materia e practical range.
|
|
|
|
|
|
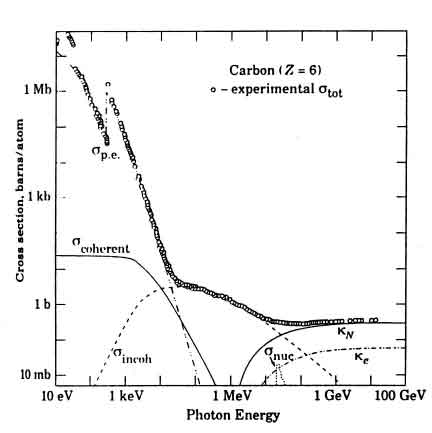
Figure 1.11: Sezione d'urto totale e contributi dei differenti processi per fotoni in carbonio, in funzione dell'energia. ( sp.e.:effetto fotoelettrico, scoherent: diffusione coerente, sincoherent: diffusione Compton, kn: produzione di coppie nel campo nucleare, ke: produzione di coppie nel campo degli elettroni, snuc: assorbimento fotonucleare )
|
|
|
|
|
|
|
|
|
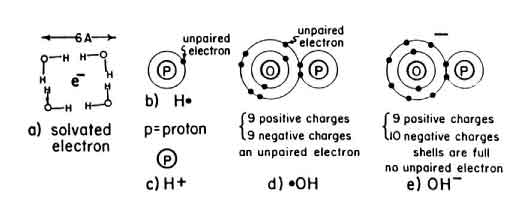
Figure 1.12: Specie reattive prodotte nella radiolisi dell'acqua: a) elettrone acquoso o solvatato eaq-, b) radicale idrogeno H�, c) ione idrogeno H+, d) radicale idrossile OH�, e) ione idrossile OH-.
|
|
|
|
|
|
|
|
|
|
|
|
|
| Particella | Carica | Energia(MeV) | LET(keV/mm) |
| Elettrone | -1 | 0.01 | 2.3 |
| 0.1 | 0.42 | ||
| 1 | 0.25 | ||
| Fotone | 0 | 1.17-1.33 | 0.2 2 |
| (g del 60Co ) | |||
| Protone | +1 | 2 | 16 |
| +2 | 8 | ||
| +5 | 4 | ||
| +10 | 0.4 | ||
| a | +2 | 5 | 95 |
| Neutrone | 0 | 5 | 3-30 |
| Ione carbonio C+6 | 6 | 10MeV/u | 170 |
| 250MeV/u | 14 | ||
|
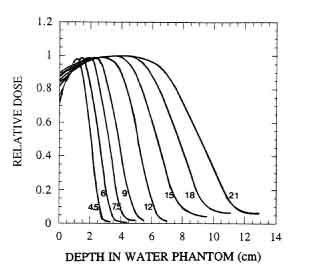
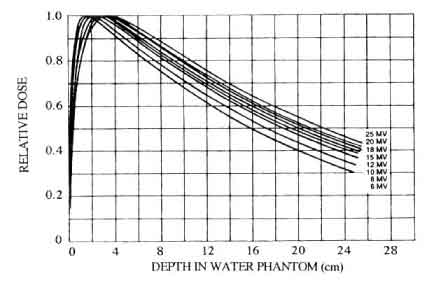
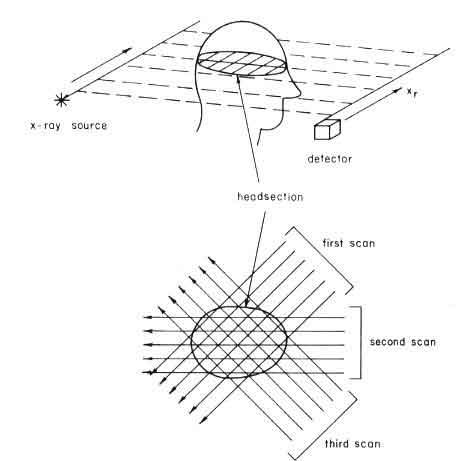
Figure 2.11: Sistema di scanning per tomografia. Un fascio di raggi X passa attraverso l'oggetto e viene rivelato all'uscita. Il sistema (sorgente di raggi X - detector) scorre lungo le direzioni indicate nella figura in alto (scansione). Piú scansioni vengono effettuate a vari angoli per poter ricostruire meglio l'immagine dell'organo.
|
|
|
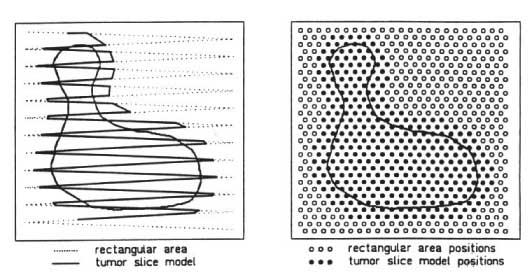
|
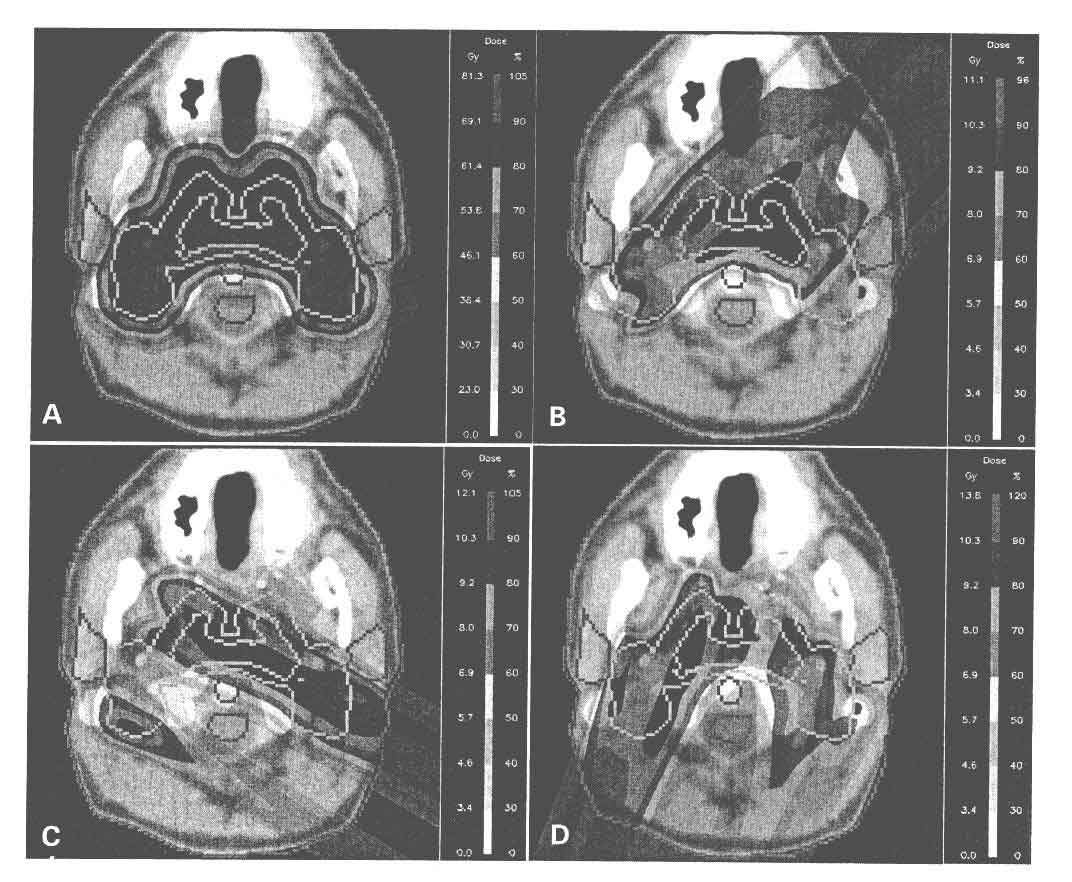

Figure 2.14: Struttura schematica di un sistema a scansione magnetica.
Dove A,B,C sono matrici.A = B + C; A = B * C;
#include <iostream.h>
int main(){
int i;
cout << ``Inserire un numero ``;
cin >> i;
cout << ``Numero inserito = '' << i;
return 0;
}
Questo programma effettua un input da tastiera (cin = console
input) ed un output su terminale (cout = console
output).
int min(int a,int b) { /* Dichiarazione */
if( a < b ) return a;
if( b < a ) return b;
}
#include <iostream.h>
int main() {
int i,j;
cout << " Inserire due numeri ";
cin >> i >> j;
cout << " Numero minore = " << min(i,j); /* Utilizzo */
return 0;
}
I file header e implementation implementazioneint min(int a,int b)
ed utilizzoif( a < b ) return a; if( b < a ) return b;
della funzione min erano tutti e tre in uno stesso file il cui nome sarebbe potuto essere ad esempio "prova.cc". Normalmente invece queste tre parti vengono suddivise in quantocout << " Numero minore = " << min(i,j);
int min(int a,int b);
#include "min.h"
int min(int a,int b){
if( a < b ) return a;
if( b < a ) return b;
}
Per ottenere il file con suffisso .o di questo programma si dovrá dare
il seguente comando:
(Si noti che con l'istruzione #include "min.h" in questo file, si hanno di fatto due dichiarazioni della funzione "min" : ció garantisce che chi scrive la implementazione non confonda qualche tipo di dato infatti se le due dichiarazioni sono uguali questo non costituisce problema ma se sono diverse la compilazione si interrompe e viene segnalato un errore.)cxx -c utilizzo.cc
#include "min.h" /* Richiamo della definizione */
#include <iostream.h>
int main() {
int i,j;
cout << " Inserire due numeri ";
cin >> i >> j;
cout << " Numero minore = " << min(i,j); /* Utilizzo */
return 0;
}
Per ottenere il file eseguibile (con suffisso .exe) di questo programma
si dovrá dare il seguente comando:
Dove l'ultimo termine (min.o) significa che viene eseguito un link (collegamento) al modulo precedentemente compilato (min.cc) in modo da poter usare la funzione min().cxx -c min.cc -o min.exe min.o
class unaClasse{
private:
int a;
public:
void set_a(int num);
int get_a();
}
file unaClasse.cc:
void unaClasse::set_a(int num)
{
a = num;
}
int unaClasse::get_a()
{
return a;
}
file utilizzo.cc:
#include "unaClasse.h"
#include <iostream.h>
int main(){
unaClasse var1;
unaClasse var2;
var1.set_a(10);
var2.set_a(22);
cout << var1.get_a();
cout << var2.get_a();
return 0
}
Questo programma, una volta compilato ed eseguito produrrá la
stampa dei numeri 10 e 22.Con queste definizione é quindi possibile usare istruzioni come:mm = 1; cm = 10 * 1 mm; m = 1000 * 1 mm; micrometer = 1.e-3 * 1 mm; nanometer = 1.e-6 * 1 mm;
oppure (usando le definizioni delle energie):thickOfDetector = 5*mm;
Tutto questo evita di dover riportare a parte le convenzioni sulle unitá di misura assunte e risulta molto chiarificativo nella lettura di codice altrui.enOfParticle = 5*MeV;
(Nella seconda istruzione non ci si cura piú delle singole componenti del puntatore a quel voxel).int3Vector spotVoxel=(10,2,13); actualDensity = densityMatrix(spotVoxel);
La prima istruzione dichiara ed inizializza la variabile spotVoxel.int3Vector spotVoxel=(10,2,13); int3Vector originIndex;
Produrrá sul video la scritta:int3Vector spotVoxel=(10,1,13); cout << spotVoxel;
[10,1,13]
int3Vector spotVoxel=(10,1,13);
cout << ``La terza componente del vettore spotVoxel vale ''
<< spotVoxel.k();
Produrrá sul video la scritta:
La terza componente del vettore spotVoxel vale 13
Produrrá sul video le scritte:int3Vector spotVoxel=(10,1,13); cout << spotVoxel << endl; spotVoxel.setK(22); cout << spotVoxel<< endl;
[10,1,13] [10,1,22]
Queste istruzioni settano una variabile logica a seconda della uguaglianza o meno dei due int3Vector. (Due int3Vector vengono ovviamente definiti uguali se hanno tutte e tre le componenti uguali)./* (actualSpot e spotVoxel sono due int3Vector) */ flagEqual = false; if (actualSpot=spotVoxel) flagEqual = true;
Hep3Vector spotPosition(111.1,22.3,313.13); Hep3Vector oldSpotPosition=spotPosition;
Produrrá sul video la scritta:int3Vector spotVoxel=(7.1,5.2,2.3); cout << spotVoxel;
(7.1,5.2,2.3)
int3Vector A=(0.0,0.0,0.0);
int3Vector B=(3.0,0.0,0.0);
cout << "La distanza tra "<< A << " e "
<< B << " vale " << A.distance(b);
Queste produrranno sul video la scritta:
La distanza tra (0.0,0.0,0.0) e (1.0,1.0,1.0) vale 1.0
#include "Line.h"
#include "SystemOfUnits"
#include "Hep3Vector"
int main(){
Hep3Vector p1(0.2*mm,0.3*mm,0.4*mm);
Hep3Vector p0(1*m,2*m,3*m);
Line beam(p1,p0);
}
Questo programma non compie nessuna operazione tranne quella di definire
una linea che "parte" dal punto p0 e "va" al punto p1.Produce sul video la scritta:/* (sia beam la linea dichiarata nell'esempio precedente) */ cout << "Direzione del beam: " << beam;
(Si noti che i valori delle coordinate sono stati stampati tutti in mm)Direzione del beam: from (0.2,0.3,0.4) to (1000,2000,3000)
Hep3Vector beamFrom(0.,0.,0.);
Hep3Vector beamTo(2*mm,2*mm,2*mm);
Line beam(lineTo,lineFrom);
cout << "Intersection with plane X=1 is "
<< beam.intersectionPlaneX(1.0);
cout << "Intersection with plane Y=1 is "
<< beam.intersectionPlaneX(1.0);
cout << "Intersection with plane Z=1 is "
<< beam.intersectionPlaneX(1.0);
Produrrá in output:
Intersection with plane X=1 is (1.1,1.1,1.1) Intersection with plane Y=1 is (1.1,1.1,1.1) Intersection with plane Z=1 is (1.1,1.1,1.1)
Il primo metodo ha come argomenti rispettivamento x0,y0,z0 coordinate dello spigolo "inferiore sinistro", seguono quindi tre coppie di dati che rappresentano il numero di voxel e la loro dimensione lungo ogni asse./* Costruttore 1: */ GridBox TTV(100*cm,-8*cm,-8*cm,52,3*mm,50,3*mm,49,3*mm); int3Vector PTVpositionInsideTTV(21,20,19); int3Vector PTVnumberOfVoxels(5,5,5); /* Costruttore 2: */ GridBox PTV(TTV,PTVpositionInsideTTV,PTVnumberOfVoxels);
Produce la seguente stampa:GridBox TTV(100*cm,-8*cm,-8*cm,52,3*mm,50,3*mm,49,3*mm); cout << "TOTAL VOLUME" << TTV;
Origin : (1000,-80,-80) Number of Voxels: [52,50,49] Voxel dimensions: (3,3,3)
Produrrá in stampaGridBox TTV(100*cm,-8*cm,-8*cm,52,3*mm,50,3*mm,49,3*mm); cout << "Numero di Voxel lungo Y = " << TTV.dimensions().i()
Numero di Voxel lungo Y = 50
La prima chiamata ha come argomento un dato di tipo int3Vector mentre la seconda ha come argomento tre interi.int3Vector spotINDEX(1,2,3); Hep3Vector spotPOINT; spotPOINT = TTV.centerPosition(spotINDEX); /* prima chiamata */ Hep3Vector firstSpotPOINT; firstspotPOINT = TTV.centerPosition(4,4,4);/* seconda chiamata */
GridBox TTV(100*cm,-8*cm,-8*cm,52,3*mm,50,3*mm,49,3*mm);
Hep3Vector P(100.1*cm,-8.1*cm,-8.1*cm);
if(TTV.include(P)){
cout << "Il punto P risiede nel voxel"
<< TTV.insideWhichVoxel(P)
}
Queste istruzioni produrrano la seguente stampa:
In quanto il controllo di appartenenza al GridBox é stato verificato.Il punto P risiede nel voxel [0,0,0]
Rijk densy( "x","y","z", 110,115,98);
Rijk initialDose("x","y","z", 110,115,98,1.0);
Rijk newDose=initialDose;
I primi tre argomenti costituiscono i nomi delle tre dimensioni e
sono utili qualora si decida di usare la stessa classe per memorizzare
dati sempre a tre indici ma il cui significato potrebbe essere diverso
(ad esempio r, q, f se la matrice contiene elementi la
cui geometria é quella di un settore di calotta sferica. Per la memorizzazione di una matrice le cui entrate sono state poste uguali ad 1.0 su file.Rijk matrixONE( "x","y","z" , 10 , 10 , 10, 1.0 ) \\ matrixONE.store(/user/rossi/myMatrix.Rijk) \\
Ripristina la matrice da file. Questo meccanismo puó essere utile ad esempio per memorizzare matrici la cui elaborazione é stata dispendiosa e sará sempre la stessa, anziché ricalcolarla ogni volta quindi la si memorizza una volta calcolata e si riutilizza in seguito il risultato ottenuto. Si consiglia di usare file con un suffisso "Rijk" per identificarne piú facilmente i contenuti. La matrice viene memorizzata nel file in ASCII (cioé con caratteri alfabetici) ordinata per slide e con corrispondenza di righe e colonne rispettivamente col primo e secondo indice (i e j). Tale scelta, coadiuvata con un editor capace di gestire linee senza limiti di lunghezza (come xemacs ) o con un sistema di stampa a molte colonne come a2ps [21] , permette un confronto diretto di una immagine di CT con i valori numerici letti (slide per slide). Ad esempio una matrice le cui entrate sono:Rijk A=Rijk_restore(/user/rossi/myMatrix.Rijk) \\
i=0 113 123 133 143
i=1 213 223 233 243 (k=2)
i=2 313 323 333 343
i=0 112 122 132 142
i=1 212 222 232 242 (k=1)
i=2 312 322 332 342
i=0 111 121 131 141
i=1 211 221 231 241 (k=0)
i=2 311 321 331 341
0 1 2 3
/ / / /
j j j j
Verrá memorizzata su file nel seguente modo:
file mat.Rijk
I dati sono scritti in ASCII con ulteriori scritte aggiunte che consentano di darne una interpretazione anche per chi eventualmente volesse leggerli ad esempio da un programma scritto in FORTRAN. Nell'ordine il significato dei dati e delle scritte é:i j k 3 4 3 dataInThisFileAreInKIJorderKmovesSlowerJFaster k=0############################## 111 121 131 141 211 221 231 241 311 321 331 341 k=1############################## 112 122 132 142 212 222 232 242 312 322 332 342 k=2############################## 113 123 133 143 213 223 233 243 313 323 333 343
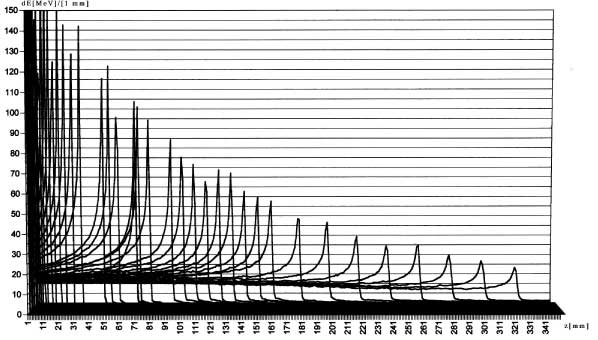
R1R1 range("Range[mm]","Energy[MeV/u]",47);
R1R1 range("Range[mm]","Energy[MeV/u]",2);
range.put(0, 47.0*mm,143.0*MeV);
range.put(1, 51.0*mm,150.0*MeV);
R1R1 range("Range[mm]","Energy[MeV/u]",2);
range.put(0, 47.3*mm,143.0*MeV);
range.put(1, 51.7*mm,150.0*MeV);
range.put(2, 56.2*mm,159.0*MeV);
range.put(3, 68.1*mm,177.0*MeV);
cout << range;
Produce la seguente stampa:
Range[mm] Energy[Mev/u] 47.3 143.0 51.7 150.0 56.2 159.0 68.1 177.0
Dove la prima riga contiene nell'ordine:Range[mm] Energy[Mev]/u 26 1 22 2 27 3 33 4 41 5 45 6 50 8 56 10 62 12 69 15 76 18 85 22 94 27 105 32 116 47 143 51 150 56 159 68 177 70 180 77 190 92 210 99 220 107 230 115 240 124 250 132 260
first first 1 2 3 (nome= "Esponente")
col row
2 2 4 8
3 3 9 27
(nome=
"PotenzeCalcolate")
5 5 25 125
10 10 100 1000
(nome=
"base")
La prima riga viene indicata come firstRow e le sue entrate
valgono rispettivamente 1, 2 e 3. Il suo nome é "Esponente".
RiRjR1 EnergyLoss("z[mm]", "En[MeV/u]", "En[MeV/ione]",351,47)
RiRjR1 EnergyLoss2=RiRjR1_restore("EnlossFromGeant.RiRjR1");
La prima dichiarazione predispone la struttura nella quale memorizzare i
dati (inizializzando a zero tutte le entrate della matrice). A tale metodo
seguirá un qualche algoritmo o simulazione per riempire con i valori
desiderati tali entrate per poi eventualmente memorizzarli su un file
con il metodo store che verrá illustrato in seguito.
#include "RiRjR1.h"
main() {
RiRjR1 tabellaEsempio("Base",
"Esponente",
"PotenzeCalcolate",
4,3)
tabellaEsempio.putFirstRow(0,1.);
tabellaEsempio.putFirstRow(1,2.);
tabellaEsempio.putFirstRow(2,3.);
tabellaEsempio.putFirstCol(0,2.);
tabellaEsempio.putFirstCol(1,3.);
tabellaEsempio.putFirstCol(2,5.);
tabellaEsempio.putFirstCol(3,10.);
tabellaEsempio.put(0,0,2.);
tabellaEsempio.put(0,1,4.);
tabellaEsempio.put(0,2,8.);
tabellaEsempio.put(1,0,3.);
tabellaEsempio.put(1,1,9.);
tabellaEsempio.put(1,2,27.);
tabellaEsempio.put(2,0,5.);
tabellaEsempio.put(2,1,25.);
tabellaEsempio.put(2,2,125.);
tabellaEsempio.put(3,0,10.);
tabellaEsempio.put(3,1,100.);
tabellaEsempio.put(3,1,1000.);
tabellaEsempio.store("TabellaEs.RiRjR1")
}
(Si noti come in C++ si usa la convenzione di far partire gli indici
da zero anziché da uno come ad esempio in FORTRAN; tale convenzione
é giustificata dall'utilizzo dei puntatori per la gestione delle
matrici o dei vettori: la prima componente di un vettore é quella che
si trova all'indirizzo di inizio del vettore, la terza quella che si trova
due locazioni dopo per questo motivo l'indice del vettore rappresenta
direttamente la dislocazione rispetto all'indirizzo di partenza solo se viene
fatto partire, appunto, da zero.)
#include "RiRjR1.h"
#include <iostream.h>
main(){
RiRjR1 tabella=RiRjR1_Restore("TabellaEs.RiRjR1")
cout << "Elemento recuperato = " << tabella.get(0,2);
}
Queste istruzioni produrranno la seguente stampa:
(Anche qui bisogna porre attenzione al fatto che gli indici 0,2 significano primo e terzo elemento.)Elemento recuperato = 8
Ovviamente ad ognuna delle parole chiavi a sinistra corrispondono uno o piú punti del programma che la richiamano con una sintassi consentita da una particolare struttura dati definita "Map" che consente di usare una stringa come se fosse un indice di un vettore. Tale vettore é unico per ogni tipo di dati: ( files__ � Nomi di Files)% Files per l'esecuzione e per i risultati: F CT_input /usr/ater/rossi/data/candiolo/73843748 F energyLoss enLoss.dat F graficoDosi dosiProva22.hbook F TPoutputFile TP22.dat % % Cose da fare o da non fare durante questa esecuzione: B debugON false B printRunSetting true % % Interi I numeroIterazioni 1000 I tipoCT 1 % (1=MOLINETTE,2= PSI, 100=GSI) % Stringhe S titoloGraficoDosi Prova22 % % Reali R XofVertex 896.0 R YofVertex 8.0 R ZofVertex -2000 % (queste lunghezze sono espresse in millimetri.)
#include "cRUN.h"
main(){
cRUN thisRun;
readDataCards("Prova1.run");
cout << files__["TPoutputFile"]
cout << files__["graficoDosi"] ;
cout << files__["energyLoss"]
cout << files__["CT_input"] ;
cout << flags__["printRunSetting"] ;
cout << flags__["debugON"]
cout << intVar__["numeroIterazioni"]
cout << intVar__["tipoCT"] ;
cout << strVar__["titoloGraficoDosi"]
cout << realVar__["ZofVertex"] ;
cout << realVar__["YofVertex"] ;
cout << realVar__["ZofVertex"] ;
}
Produce in stampa:
TP22.dat
dosiProva22.hbook
enLoss.dat
/usr/ater/rossi/data/candiolo/73843748
true
false
1000
1
Prova22
-2000
8.0
896.0
(L'ordine di alcuni dati é stato volutamente cambiato rispetto a quello
del file di input
per indicare
che essi possono essere richiamati in qualsiasi ordine all'interno
del programma).
Questo programma é stato eseguito con i dati impostati nel file Prova1.run ma
qualora anziché apportare continue modifiche al file Prova1.run si decida
di avere predisposti piú file di esecuzione ( Prova1.run, Prova2.run,etc.)
il nome del file di esecuzione potrebbe essere
variabile e passato in input
da tastiera prima della istruzione readDataCards():
String nomeFileEsecuzione; cin >> nomeFileEsecuzione; cRUN thisRun=readDataCards(nomeFileEsecuzione);
Crea sei mappe:cRUN thisRun;
cRUN thisRun;
thisRun.declareFile("CTinput", "23322343.dat");
cRUN thisRun;
/*1*/ thisRun.declareFile("CTinput", "23322343.dat");
/*2*/ thisRun.declareFlag("debugON", false);
/*3*/ thisRun.declareIntVar("tipoCT", 1);
/*4*/ thisRun.declareStrFile("titoloGraficoDosi", "prova22");
/*5*/ thisRun.declareRealFile("XofVertex", 89.6*cm);
#include ``cRUN.H''
main(){
cRUN thisRun;
readDataCards("Prova1.run");
/*1*/ cout << files__["TPoutputFile"]
/*2*/ cout << flags__["printRunSetting"] ;
/*3*/ cout << intVar__["numeroIterazioni"]
/*4*/ cout << strVar__["titoloGraficoDosi"]
/*5*/ cout << realVar__["ZofVertex"] ;
}
I risultati di queste stampe sono giá stati presentati negli esempi
precedenti.Le righe A1-A3 indicano che al comando dato da tastiera "gmake useR1R1" il computer deve eseguire l'istruzione "useR1R1.exe" a condizione che "useR1R1.exe" (dipendenza di "useR1R1") sia "aggiornato".# (Linee precedute dal carattere diesis sono COMMENTI) # (righe A1 A2 A3) useR1R1: useR1R1.exe (tab) echo running useR1R1 (tab) useR1R1.exe # (righe B1 B2 B3) useR1R1.exe: useR1R1.o R1R1.o range.o (tab) echo linkink useR1R1 (tab) cxx -o useR1R1.exe useR1R1.o R1R1.o range.o # (righe C1 C2 C3) useR1R1.o: useR1R1.cc (tab) echo compiling useR1R1.cc (tab) cxx -c useR1R1.cc # (righe D1 D2 D3) R1R1.o: R1R1.cc R1R1.h (tab) echo compiling R1R1.cc (tab) cxx -c R1R1.cc # (righe E1 E2 E3) range.o: range.cc range.h (tab) echo compiling range.cc (tab) cxx -c range.cc
|
|
|
Le seguenti tre istruzioni all'inizio del programma:
RiRjR1 enLoss = Rijk_restore("/user/ater/nicco/data/enLoss.RiRjR1");
R1R1 rangeEn= R1R1_restore("/user/ater/nicco/data/rangeEn.RiRjR1");
Rijk enLoss =RiRjR1_restore("/user/ater/nicco/data/psiDensy.Rijk");
leggono rispettivamente:
open(unit=42,FILE='psiDensy.Rijk',status='unknown')
write(42,*) 'i ','j ','k '
write(42,*) NPIX,NPIY,NPIZ
write(42,*) 'dataInThisFileAreInKIJorderKmovesSlowerJFaster'
do k=1,NPIZ
write(42,*) 'k=',k,'###################################'
do i=1,NPIX
write(42,900)( densy(i,j,k),j=1,NPIY)
end do
end do
close(42)
900 format(100(f6.3,1x))
ed il file prodotto costituisce la sorgente CT sulla quale é stato testato il
programma.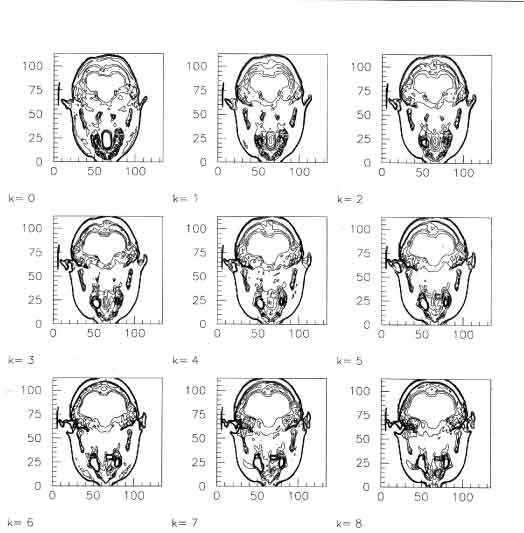
GridBox TTV( 0*cm,0 *cm, 0*cm, 134,1.594*mm ,113,1.594*mm ,98,1.594*mm ); int3Vector TGVposInsideTTV(40,40,40); int3Vector dimOfTGV(20,20,2); Hep3Vector source(-400*cm,8.61*cm,7.97*cm);La prima istruzione indica al programma che il volume della CT (ToTal Volume) é posizionato al centro del sistema di riferimento adottato (0*cm,0*cm,0*cm...), ha 134*113*98 voxel ognuno di dimensione 1.594*1.594*1.549 mm.
cout<<"(All lenghts are in mm)"<< endl;
cout<<"TTV:"<< TTV << endl;
cout<< "Lower & Upper limits:"<< TTV.lowerCorner()<< " "
<<TTV.higherCorner()<<endl;
cout<<"TGV:"<< TGV << endl;
cout<< "Lower & Upper limits:"<< TGV.lowerCorner()<< " "
<<TVV.higherCorner()<<endl;
cout<<"Source:" << source << endl;\
e producono il seguente risultato:
(All lenghts are in mm) TTV: Origin : (0,0,0) Number of Voxels: [134,113,98] Voxel dimensions: (1.594,1.594,1.594) Lower & Upper limits:(0,0,0) (213.596,180.122,156.212) TGV: Origin : (63.76,63.76,63.76) Number of Voxels: [10,10,10] Voxel dimensions: (1.594,1.594,1.594) Lower & Upper limits:(63.76,63.76,63.76) (79.7,79.7,79.7) Source:(-4000,86.1,79.7)
for( iz=0; iz<TGV.nVoxels().k();iz++){
for( iy=0; iy<TGV.nVoxels().j();iy++){
for( ix=0; ix< TGV.nVoxels().i() ;ix++){
...
...
...
}
}
}
IX,IY,IZ sono gli indici del voxel sul quale é puntato lo spot rispetto al volume CT (TTV), spotInd é la versione vettore dei suddetti tre indici, spot é la posizione nello spazio del centro del voxel e beam é la linea che parte dalla sorgente (source) e passa attraverso il centro di quel voxel (spot).IX=ix+TGVposInsideTTV.i(); IY=iy+TGVposInsideTTV.j(); IZ=iz+TGVposInsideTTV.k(); spotInd = int3Vector(IX,IY,IZ); spot = TTV.centerPosition(spotInd); Line beam(source,spot);
BeamOrthogonalToX=false;
BeamOrthogonalToY=false;
BeamOrthogonalToZ=false;
if(abs((source.x()-spot.x()))< ParallelismLimit)BeamOrthogonalToX=true;
if(abs((source.y()-spot.y()))< ParallelismLimit)BeamOrthogonalToY=true;
if(abs((source.z()-spot.z()))< ParallelismLimit)BeamOrthogonalToZ=true;
if(!BeamOrthogonalToX){
for(double x=TTV.lowerCorner().x();
x<=TTV.higherCorner().x();
x+=TTV.voxDims().x() )
{
tempPoint= beam.intersectionPlaneX(x);
if(TTV.include(tempPoint))
{
vect_distUintersPnt[iInters].dist=source.distance(tempPoint);
vect_distUintersPnt[iInters].intersPnt=tempPoint;
iInters++;
}
}
}
if(!BeamOrthogonalToY){....}
if(!BeamOrthogonalToZ){....}
Le prime sei istruzioni servono ad accertarsi che vi siano intersezioni:
ParallelismLimit é un numero molto piccolo che garantisce che intersezioni
piú distanti di 1010 mm non vengano neanche trattate, altrimenti il
calcolatore andrebbe in errore da "overflow".sort(vect_distUintersPnt.begin(),&vect\_distUintersPnt[iInters-1])Ordina il vettore vect_distUintersPnt sulla base di un criterio fornito in precedenza:
bool operator <(const distUintersPnt &x,const distUintersPnt &y)
{
return x.dist < y.dist ;
}
cioé sulla base della distanza.#include <vector> #include <algorithm> #include <functional>
struct distUintersPnt{
double dist;
Hep3Vector intersPnt;
};
vector<distUintersPnt> vect_distUintersPnt(NmaxIntersections);
bool operator <(const distUintersPnt &x,const distUintersPnt &y)
{
return x.dist < y.dist ;
}
sort(vect_distUintersPnt.begin(),&vect_distUintersPnt[iInters-1]);
for(int ii=0;ii<nInters-1;ii++)
{
middlePnt=(vect_distUintersPnt[ii].intersPnt+
vect_distUintersPnt[ii+1].intersPnt) *0.5;
vintersInd[ii]=TTV.insideWhichVoxel(middlePnt);
vpath[ii]=vect_distUintersPnt[ii].intersPnt.
distance(vect_distUintersPnt[ii+1].intersPnt);
vWEpath[ii]=vpath[ii]*densy.get(vintersInd[ii]);
WEpathTot+=vWEpath[ii];
vWEpathTot[ii]=WEpathTot;
vWEspotCenter[ii]=vWEpathTot[ii]- vWEpath[ii]/2;
if(vintersInd[ii]==spotInd)iSPOT=ii;
}
L'ultima istruzione serve a memorizzare in quale posizione sequenziale
si trova il voxel verso il quale era diretto lo spot
(una volta che le intersezioni sono state ordinate).
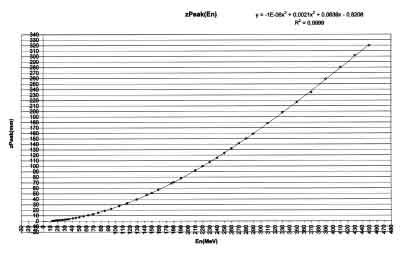
E' quindi possibile calcolare l'energia necessaria al fine di avere un picco di Bragg in quella posizione:zpeak=vWEspotCenter[iSPOT];
Occorre adesso stabilire quanta energia rilascerá in ogni voxel una particella avente energia E al fine di poter calcolare le fluenze.E=rangeEn.linInterpolation(zpeak);
for(int ii=0; ii<nInters-1;ii++){
dEvox.put(iBEAM,ii,(V[ii]*MeVtoJoule)/
(voxVolume*densy.get(vintersInd[ii])*10E-6) );
}
\\ 1 gm/cm3 = 10^(-3)kg/10^(3)mm3 --> *10E-6
Nel terzo argomento del metodo "put" viene realizzata la conversione
da MeV/voxel a Gray: J/kg.
Alla fine del ciclo sulla "fila" dEvox (dichiarata di tipo RiRjR1)
conterrá il rilascio di dose sopra indicato.
Producono la seguente stampa:cout << "Beam = " << beam << endl; cout << "spotInd " << spotInd << endl; cout << "spotPos " << spot << endl; cout << "zpeak = " << zpeak << endl; cout << "En = " << En << "MeV" <<endl;
Inoltre é possibile stampare le informazioni relative a tutti i voxel attraversati:Beam = from (-4000,86.1,79.7) to (64.557,64.557,64.557) spotInd [40,40,40] spotPos (64.557,64.557,64.557) zpeak = 24.5792 En = 98.5742 MeV
for(int ii=0; ii<nInters-1;ii++){
cout << setw(4) << ii
<< " " << vect_distUintersPnt[ii].intersPnt
<< " " << setw(10) << vect_distUintersPnt[ii].dist
<< " " << setw(10) << vpath[ii]
<< " " << vintersInd[ii]
<< " " << setw(10) << densy.get(vintersInd[ii])
<< " " << setw(10) << vWEpath[ii]
<< " " << setw(10) << vWEpathTot[ii]
<< " " << setw(10) << D[ii]
<< " " << setw(10) << D[ii]*MeVtoJoule
<< " " << setw(10) << dEvox.get(iBEAM,ii)
<< endl;
}
Intestazione a parte, le precedenti istruzioni producono la stampa in
fig.
4.4
e fig.
4.5 .
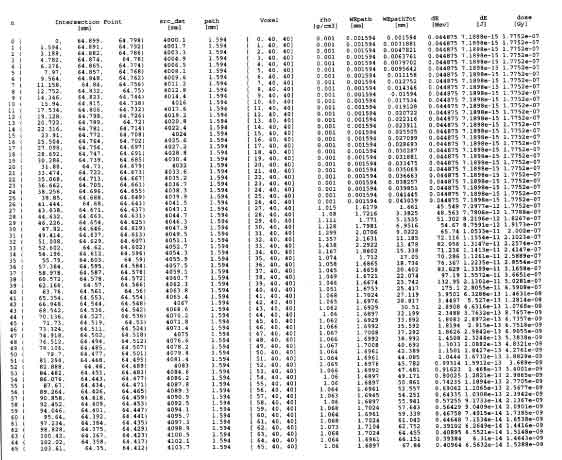
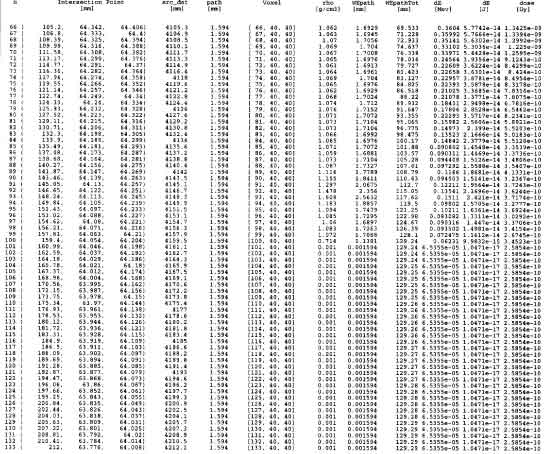
R1R1 fluence = findFluence(dEvox,TGVposInsideTTV.i(),dimOfTGV.i() ); cout << fluence<< endl;Produranno la stampa (giá intestata) della due colonne di numeri fluenza-dose della quale vengono qui riportate le sole righe dei voxel vicini al bersaglio:
0 0.59612 0 0.59622 0 0.59812 0 0.6035 0 0.61325 0 0.6284 0 0.65186 0 0.67924 0 0.70519 0 0.73203 0 0.75916 0 0.79259 0 0.83806 0 0.90455 1.5767e+05 1 1.6444e+05 1 1.963e+05 1 1.9463e+05 1 2.7161e+05 1 2.8179e+05 1 2.9948e+05 1 3.8605e+05 1 4.7864e+05 1 1.4235e+06 1 0 0.1206 0 0.030101 0 0.02549 0 0.021223 0 0.018836 0 0.016561 0 0.015258 0 0.014262 0 0.012307 0 0.011043 0 0.010058 0 0.0091796 0 0.0084665Si puó notare (fig. 4.6)
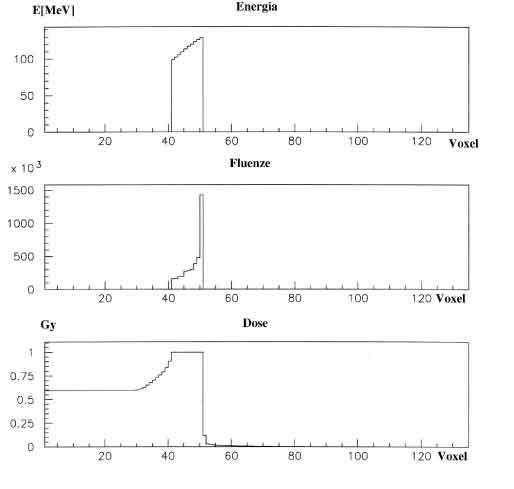
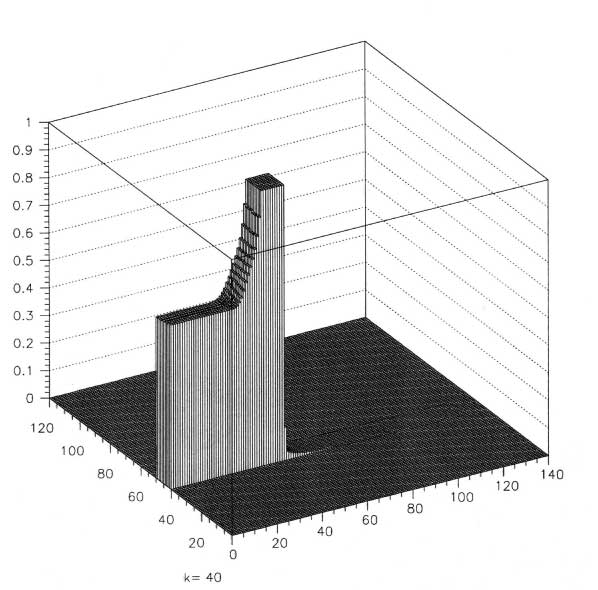
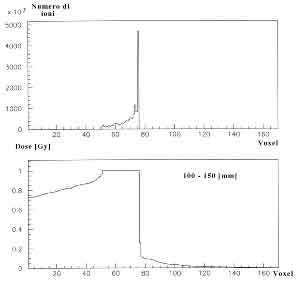
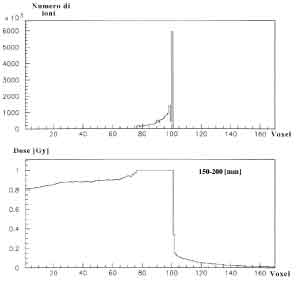
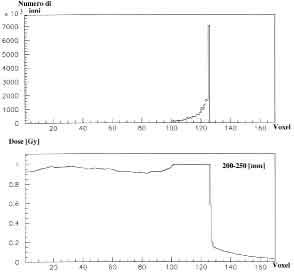
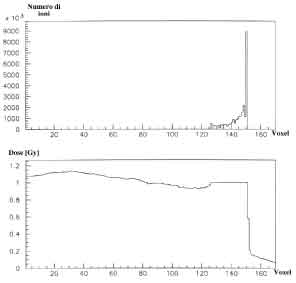
Figure 4.11: Fluenze e dosi calcolate per un SOBP in acqua realizzato con ioni carbonio alla profonditá di 250-300 mm.
| Offset | Parametro | Significato | Tipo |
| 0 | FYLTYP | identificativo del tipo di file = 6 | Int 16 bit |
| 2 | NSLICE | numero di slices nel file = 1 | Int 16 bit |
| 4 | LENGHT | numero totale di records | Int 16 bit |
| 6 | STBLK | offset del primo record contenente dati | Int 16 bit |
| 8 | FLDATE | data creazione file=anno*512+mese*32+giorno | Int 16 bit |
| 10 | IMORGN | disp. diagn.:1000-1999=CT;-2999=NMR;-3999PET | Int 16 bit |
| 12 | ZPOS | posizione relativa della slice in mm | Int 16 bit |
| 14 | NX | dim. x della matrice = 256 | Int 16 bit |
| 16 | NY | dim. y della matrice = 256 | Int 16 bit |
| 18 | XPXSZE | dimensione fisica del pixel lungo x (*1000) | Int 16 bit |
| 20 | YPXSZE | dimensione fisica del pixel lungo y (*1000) | Int 16 bit |
| 22 | DX | num. righe NON memorizzate | Int 16 bit |
| 24 | DY | num. colonne NON memorizzate | Int 16 bit |
| 26 | STATUS | 0 se ROI non memorizzato, 1 viceversa | Int 16 bit |
| 28 | free | Int 16 bit | |
| 92 | ROIBIT | marker se ROI memorizzato | Int 16 bit |
| 108 | ORIENT | orien.slice(0-4):ignoto,transv.,cor.,sag.,portfilm | Int 16 bit |
| 110 | PATORI | orientamento del paziente | Int 16 bit |
| 112 | free | Int 16 bit | |
| 140 | SEQNO | numero di sequenza interno alla CT | Char |
| 152 | SCDATE | data della scansione | Char |
| 164 | CTTYP | tipo dispositivo diagnostico | Char |
| 204 | HSPNAM | nome della clinica/ospedale | Char |
| 244 | PATID | identificativo del paziente | Char |
| 260 | PATNAM | nome del paziente | Char |
| 300 | CMNTS1 | commenti 1 | Char |
| 340 | CMNTS2 | commenti 2 | Char |
| 380 | CMNTS3 | commenti 3 | Char |
| 420 | CMNTS4 | commenti 4 | Char |
| 460 | REX | coordinata x per slice saggitale (cm) | Real 32 bit |
| 464 | REY | coordinata y per slice coronale (cm) | Real 32 bit |
| 468 | RPX | REX in pixel | Int 16 bit |
| 470 | RPY | REY in pixel | Int 16 bit |
| 472 | ISET_Z | 1 se REZ ha dati validi | Int 16 bit |
| 474 | free | Int 16 bit | |
| 476 | REZ | coordinata z nel pixel centrale (riga 128) | Real 32 bit |
| 480 | free | Int 16 bit | |
| Offset | Parametro | Significato | Tipo |
| 0 | nPNTS[10] | Numero di pnti in ogni contorno 6 | Int 8 bit |
| 10 | CMTS1 | Commenti | Char |
| 41 | IDSPWW | Larghezza finestra immagine | Int 8 bit |
| 42 | IDSPWL | Posizione finestra immagine | Int 8 bit |
| 44 | CMTS2 | Commenti | Char |
|
|
|
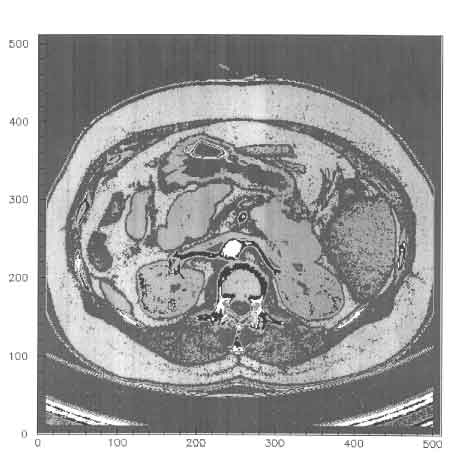
http://idt.net/~dclunie/dicom-status/status.html#BaseStandard1999si trovano tutti i formati dei dati trasmessi (spiegati dal record fino al bit) e la classificazione dei data elements. Qualora per qualche motivo il sito non fosse accessibile si riesce comunque abba stanza in fretta convergere verso questa documentazione con un motore di ricerca quale www.google.com ricercando "DICOM" (sono tutti file PDF leggibili sotto unix con il comando "gv" o sotto Windows o Mac tramite pluggin "Acrobat Reader" scaricabile da rete )
Posizione del |TAG| Lunghezza byte --> Dato associato dal 'DICOM BROWSER' realizzato in C++ (non sono presenti tutti) 00] | 0| 8| 0| 0| ( 4) --> 12] | 0| 8| 0| 5| ( 10) --> 30] | 0| 8| 0| 8| ( 30) --> Image Type= ORIGINAL\PRIMARY\AXIAL\VOLUME 68] | 0| 8| 0|16| ( 26) --> SOP Class UID= 1.2.840.10008.5.1.4.1.1.2 102] | 0| 8| 0|18| ( 48) --> SOP Instance UID= 1.3.46.670589.10.1.1.2158435922.934292566.34615. 158] | 0| 8| 0|20| ( 8) --> Study Date= 19990810 174] | 0| 8| 0|21| ( 8) --> 190] | 0| 8| 0|23| ( 8) --> Image Date= 19990810 206] | 0| 8| 0|30| ( 14) --> Study Time= 151329.234768 228] | 0| 8| 0|31| ( 14) --> 250] | 0| 8| 0|33| ( 14) --> Image Time= 154139.000000 272] | 0| 8| 0|50| ( 0) --> Accession Number= 280] | 0| 8| 0|60| ( 2) --> Modality= CT 290] | 0| 8| 0|70| ( 24) --> Manufacturer= Philips Medical Systems 322] | 0| 8| 0|80| ( 10) --> Institution Name= Philips CT 340] | 0| 8| 0|90| ( 0) --> Referring Physician's Name= 348] | 0| 8|10|10| ( 2) --> Station Name= 358] | 0| 8|10|30| ( 2) --> Study Description= 368] | 0| 8|10|40| ( 10) --> 386] | 0| 8|10|50| ( 0) --> 394] | 0| 8|10|60| ( 0) --> 402] | 0| 8|10|90| ( 12) --> Manufacturer's Model Name= Tomoscan CS 422] | 0| 8|11|40| (110) --> 540] | 0|10| 0| 0| ( 4) --> 552] | 0|10| 0|10| ( 22) --> Patient's Name= CT Aura-Secura^Example 582] | 0|10| 0|20| ( 14) --> Patient ID= ID200631zw609 604] | 0|10| 0|30| ( 8) --> Patient's Birth Date= 19991213 620] | 0|10| 0|40| ( 2) --> Patient's Sex= M 630] | 0|18| 0| 0| ( 4) --> "Calling ghostscript to convert zImages/dic1.EPSF to zImages/dic1.EPSF.gif , please wait ..." 642] | 0|18| 0|10| ( 4) --> Contrast/Bolus Agent= NONE 654] | 0|18| 0|50| ( 8) --> Slice Thickness= 5.000000 670] | 0|18| 0|60| ( 10) --> KVP= 120.000000 688] | 0|18|10|20| ( 18) --> Software Version(s)= CT Backend R1.1L1 714] | 0|18|10|30| ( 22) --> 744] | 0|18|11| 0| ( 10) --> 762] | 0|18|11|20| ( 8) --> Gantry/Detector Tilt= 0.000000 778] | 0|18|11|30| ( 10) --> Table Height= 38.000000 796] | 0|18|11|50| ( 4) --> Exposure Time= 1000 808] | 0|18|11|51| ( 4) --> 820] | 0|18|11|52| ( 4) --> Exposure= 100 832] | 0|18|12| 0| ( 8) --> Date of Last Calibration= 19990810 848] | 0|18|12| 1| ( 14) --> Time of Last Calibration= 083759.582871 870] | 0|18|12|10| ( 4) --> Convolution Kernel= AA0 882] | 0|18|51| 0| ( 4) --> Patient Position= HFS 894] | 0|20| 0| 0| ( 4) --> 906] | 0|20| 0| D| ( 48) --> Study Instance= 1.3.46.670589.10.1.1.2158435922.934290809.23985. 962] | 0|20| 0| E| ( 48) --> Series Instance= 1.3.46.670589.10.1.1.2158435922.934292556.78163. 1018] | 0|20| 0|10| ( 0) --> Study ID= 1026] | 0|20| 0|11| ( 2) --> Series Number= 19 1036] | 0|20| 0|12| ( 2) --> Acquisition Number= 1 1046] | 0|20| 0|13| ( 2) --> Instance Number= 1 1056] | 0|20| 0|32| ( 42) --> Image Position (Patient)= -2.042221e+02\-1.818909e+02\-1.570000e+02 1106] | 0|20| 0|37| ( 78) --> Image Orientation (Patient)= 1.000000e+00\0.000000e+00\0.000000e+00\0.00000.. 1192] | 0|20| 0|52| ( 48) --> Frame of Reference= 1.3.46.670589.10.1.1.2158435922.934292075.37016. 1248] | 0|20|10|40| ( 0) --> Position Reference Indicator= 1256] | 0|20|10|41| ( 12) --> 1276] | 0|20|40| 0| ( 0) --> Image Comments= 1284] | 0|28| 0| 0| ( 4) --> 1296] | 0|28| 0| 2| ( 2) --> Samples per Pixel= 1306] | 0|28| 0| 4| ( 12) --> Photometric Interpretation= MONOCHROME2 1326] | 0|28| 0|10| ( 2) --> Rows= 512 1336] | 0|28| 0|11| ( 2) --> Columns= 512 1346] | 0|28| 0|30| ( 26) --> Pixel Spacing= 7.105113e-01\7.105113e-01 1380] | 0|28| 1| 0| ( 2) --> Bits Allocated= 1390] | 0|28| 1| 1| ( 2) --> Bits Stored= 1400] | 0|28| 1| 2| ( 2) --> High Bit= 1410] | 0|28| 1| 3| ( 2) --> Pixel Representation= 1420] | 0|28|10|50| ( 26) --> Window Center= 4.000000e+01\7.000000e+01 1454] | 0|28|10|51| ( 26) --> Window Width= 4.000000e+02\2.000000e+02 1488] | 0|28|10|52| ( 12) --> Rescale Intercept= -1200.000000 1508] | 0|28|10|53| ( 8) --> Rescale Slope= 1.000000 1524] |7F|E0| 0| 0| ( 4) --> 1536] |7F|E0| 0|10|(524288)--> Pixel Data=